La réalité virtuelle au laboratoire de Biochimie Théorique - toucher, déformer et assembler des molécules du bout des doigts
Ces dix dernières années, la société a été le témoin de progrès importants en physique et en chimie de l’infiniment petit. Nos recherches dans ce domaine portent sur le développement et l’utilisation de simulations numériques pour l’étude des biomolécules. Ces simulations miment par exemple le comportement dynamique extrêmement complexe de molécules comme l’ADN, support de l’information génétique, ou comme les protéines, à la fois ouvriers et briques de la cellule. Ces macromolécules biologiques assurent leur fonction en interagissant sélectivement entre elles au cours d’un gigantesque « LEGO™ » moléculaire au cœur de l’usine cellulaire.
Pour explorer ce LEGO moléculaire, nous développons une approche pluridisciplinaire combinant les simulations moléculaires et les techniques issues de la réalité virtuelle. Dans cette approche, le chercheur peut observer en temps réel les mouvements qui animent les molécules durant la simulation. Il peut aussi saisir, allonger et manipuler ces molécules de manière interactive, pour étudier à la fois leurs déformations et leurs agencements, dont la compréhension est primordiale pour mettre en lumière les dysfonctionnements à l’échelle moléculaire qui donnent naissance à des maladies. Ceci est facilité par une manipulation très intuitive des molécules qui sont rendues palpables par un périphérique à retour d’effort. Nous avons déjà appliqué cette méthode à de nombreuses macrostructures moléculaires, notamment des groupes de protéines impliquées dans la fusion membranaire, avec des résultats scientifiques prometteurs. Nous cherchons à avancer dans la compréhension de diverses maladies comme le botulisme ou le tabagisme, ou encore à comprendre des processus fondamentaux comme la réparation de l’ADN endommagé dans la cellule.
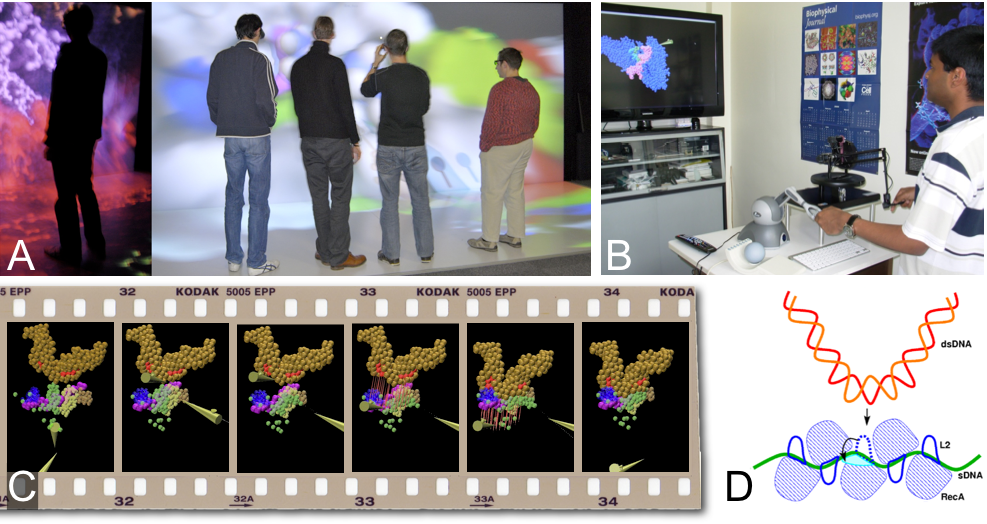
A- Séance de travail sur la plateforme Holo3 (photos © Z. Lu). B- arrimage interactif de l’ADN sur le nucléofilament RecA. C- séquence d’images de la manipulation. D- Vue schématique de la tâche à accomplir. Des détails sur B-D sont publiés dans Nucleic Acids Res. 38, 2010, 6313-23.
Un premier résultat marquant est le travail accompli pour rendre nos logiciels performants, étape indispensable pour une utilisation interactive de ceux-ci. Nous travaillons en ce moment sur l’amélioration de différentes boucles de simulation, de visualisation et d’interaction coûteuses en calcul. En utilisant l’intergiciel FlowVR, il est possible de contrôler finement ces éléments pour des simulations interactives haute performance couplant réalité virtuelle, visualisation scientifique et simulations parallèles. Pour un couplage simple, nous avons développé l’intergiciel MDDriver et un visualiseur interactif basé sur VTK. Nous avons été amenés à développer de nouvelles représentations moléculaires, plus efficaces et plus adaptées, opérant directement sur les cartes graphiques à travers des ombreurs (shader) Cg et GLSL. Actuellement nous explorons le potentiel des moteurs de jeux (en particulier Unity3D) pour de telles applications. La multimodalité, l’interaction immersive et collaborative, la navigation, la sélection, la gestion des périphériques et l’interface utilisateur en général présentent des défis importants que nous souhaitons approfondir dans un futur proche.
Afin de mieux et plus largement faire connaître les approches de réalité virtuelle pour le calcul intensif en biologie, nous visons en premier lieu des scénarios de bureau ou de salles à faible coût d’équipement. Au sein du laboratoire nous avons dédié une pièce spécifique pour les manipulations interactives et nous disposons de bras haptiques, périphériques de navigation, écrans stéréoscopiques et stations graphiques. Des essais sur des dispositifs plus immersifs se font en collaboration avec nos partenaires. L'équipe est dirigée par M. Baaden (CR CNRS) et comporte trois doctorants (A. Tek, B. Laurent, Z. Lu) et un post-doctorant (M. Piuzzi). Elle bénéficie d'un appui régulier de M. Chavent (post-doctorant au CEA) dans le cadre du projet FlowVRNano. Des collaborations fructueuses existent avec le laboratoire d’informatique fondamentale de l’université d’Orléans (LIFO), le projet MOAIS de l’INRIA Grenoble, le CEA/DIF à Bruyères-le-Châtel, le LIMSI à Orsay, le groupe ALICE à Nancy et la plateforme HOLO3 à Strasbourg. Ce travail est soutenu par le projet ANR FlowVrNano. Plusieurs publications sont parues, entre autres dans J. Comput. Chem., Nucleic Acids Res., VRST’08, VRIPHYS’08 et Pac. Symp. Biocomputing.