Comment fonctionne la DNase I, un agent thérapeutique encore mal connu ?
L'enzyme DNase I, bien connue pour être un outil largement utilisé en biochimie et biologie moléculaire, est utilisée en médecine pour lutter contre la mucoviscidose. Des chercheurs du CNRS ont réussi à comprendre, grâce à des simulations au niveau atomique, le rôle fondamental joué par les ions magnésium et calcium dans le fonctionnement de la DNase I. Ces travaux ont donné lieu à une publication dans la revue PLOS Computational Biology le 18 novembre 2010 et ouvrent la voie à l'amélioration de médicaments à base de cette enzyme.
La DNase I est l’enzyme responsable de la digestion de l’ADN extracellulaire. Ses capacités à hydrolyser l’ADN sont couramment exploitées en biochimie, par exemple pour détecter l’empreinte des protéines fixées sur un génome cellulaire. En médecine, la forme recombinante humaine de la DNase I est, sous le nom de Pulmozyme®, le plus puissant mucolytique utilisé dans le traitement permanent et quotidien de la mucoviscidose.
Malgré sa large utilisation et surtout son importance médicale, le système DNase I/ADN était mal connu. Avant les travaux récents de trois équipes de chercheurs du CNRS, les propriétés de la protéine et de l’ADN qui permettent la formation du complexe actif étaient passablement obscures. De même, dans le complexe, la structure du site catalytique dont va dépendre le mécanisme de l’hydrolyse restait largement hypothétique. Ces questions relèvent de la recherche fondamentale, mais concernent aussi la recherche appliquée : la conception d’agents thérapeutiques plus efficaces ne peut en effet s’abstraire de ces informations.
Trois groupes de chercheurs du CNRS ont contribué à un travail qui combine simulations numériques et données expérimentales : les équipes de Marc Baaden au Laboratoire de Biochimie Théorique (LBT, CNRS/IBPC/Université Paris 7), de Brigitte Hartmann au laboratoire des Protéines de la Membrane Erythrocytaire et Homologues Non-Erythroïdes (équipe DSIMB, Inserm/INTS/Université Paris 7) et de Daniel Picot au Laboratoire de Biologie Physico-Chimique des Protéines Membranaires (LBPCPM, CNRS/Université Paris 7).
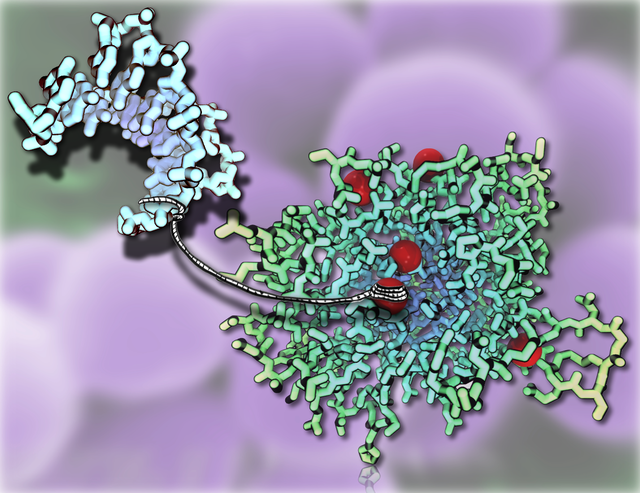
Les chercheurs ont étudié d’une part des substrats d’ADN plus ou moins bien hydrolysés par la DNase I, et d’autre part, la DNase I bovine. Ils ont ainsi montré que plus les séquences d’ADN étaient flexibles, plus elles étaient attractives pour la DNase I et donc mieux dégradées (1). Côté protéine, ils ont récemment mis en évidence cinq sites capables de fixer des cations divalents : deux cations tapissent le site catalytique et, collectivement, les cinq cations assurent la complémentarité électrostatique entre la DNase I et l’ADN, fortement chargé négativement (2).
Après avoir ainsi caractérisé les propriétés des partenaires isolés, les chercheurs vont prochainement étudier le complexe formé entre l’ADN et la DNase I. Leur but est de comprendre en détail le mécanisme catalytique, qui engage la protéine, l’ADN et deux ions. A terme, l’enjeu est de proposer des mutations qui pallieraient aux limitations de la DNase I humaine recombinante utilisée actuellement en médecine.
Figure : Des cations (en rouge) liés à la DNase I (en vert) attirent l'ADN (en bleu) pour la lier à l'enzyme. L'interface de l'enzyme est donc préorganisée par cinq sites occupés par des ions calcium et magnésium. L'image de fond représente des bactéries de staphylococcus aureus. © M. Baaden, M. J. Arduino, J. Haney Carr
Notes
- Sequence-dependent DNA flexibility mediates DNase I cleavage. J. Abi Ghanem, B. Heddi, M. Lavigne, B. Hartmann, Journal of Molecular Biology (JMB), January 8, 2010, Volume 395, Issue 1, pages 123–133.
- Ces travaux ont bénéficié d’un soutien de l'Agence Nationale de la Recherche Scientifique (Contrats ANR-06-PCVI-0025 et ANR-07-CIS7-003-01) et du Centre National de la Recherche Scientifique (Bourse de Thèse M.G.).